website links
26、
25、14种珊瑚转录组数据库(内含其他4个珊瑚转录组来源数据库)ScleractiniaTBase (hkbu.edu.hk)
1、美国国家生物信息中心National Center for Biotechnology Information (nih.gov)
2、清华大学生物信息教学Bioinformatics Tutorial - Basic - Bioinformatics Tutorial - Basic (gitbook.io)
3、 濒危物种红色名录IUCN Red List of Threatened Species
4、生物绘图在线网站BioRender
5、系统发育树在线编辑iTOL: Interactive Tree Of Life (embl.de)
6、蛋白结构域在线预测,也可下载软件进行本地批量运行Pfam: Home page (xfam.org)
7、提供的服务包括搜索、最新新闻、地图和百科、电子信箱、电子商务、互联网广告及其他服务。Yandex在俄罗斯本地的市场份额已远超俄罗斯googleYandex
8、ADW是动物自然历史百科全书,由学 ...
10个便捷的Linux小工具
Linux(1)axel多线程下载工具,下载文件时可以替代curl、wget
axel -n 20 http://centos.ustc.edu.cn/centos/7/isos/x86_64/CentOS-7-x86_64-Minimal-1511.iso
(2)shellcheckshell脚本静态检查工具,能够识别语法错误以及不规范的写法.
(3)fzf命令行下模糊搜索工具,能够交互式智能搜索并选取文件或者内容,配合终端ctrl-r历史命令搜索简直完美(ctrl + r查找以前输入的命令,比上下键和history好用)。
(4)htophtop: 提供更美观、更方便的进程监控工具,替代top命令。
(5)ag递归搜索文件内容,类似grep 和 find,但是执行效率比后两者高
参数与grep存在相似之处,如-i -A -B -C,
ag –ignore-dir :忽略某些文件目录进行搜索,
ag -w PATTERN: 全匹配搜索,只搜索与所搜内容完全匹配的文本,
ag –java PATTERN: 在java文件中搜索含PATTERN的文本,
ag –xml PATTERN ...
海洋鱼类的悲哀
那么利用率“不足100%”到底指什么呢?难道海洋中鱼类的存在仅仅是为了被人类利用吗?(Earle S A, 1995;Weber P, 1993)
Knowledge of Sequencing
浅谈测序
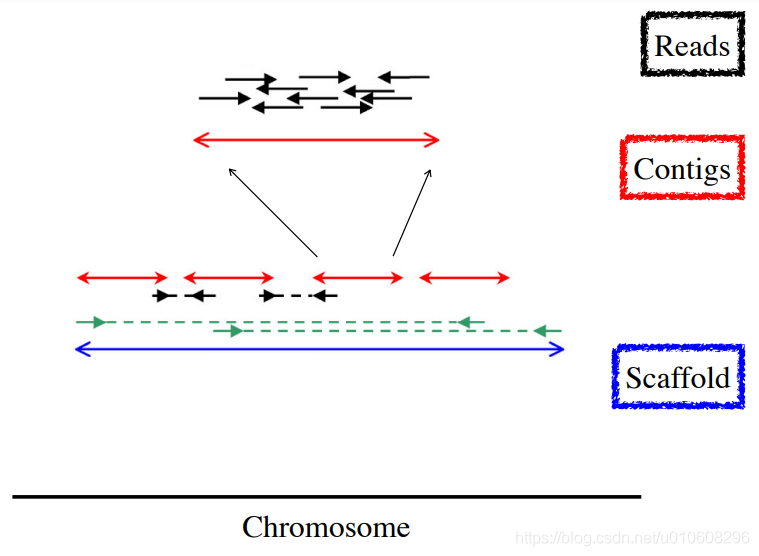
基因组组装是指使用测序方法将待测物种的基因组生成序列片段(即read),并根据reads 之间的重叠区域对片段进行拼接,先拼接成较长的连续序列(contig),再将contigs 拼接成更长的允许包含空白序列(gap)的scaffolds,通过消除scaffolds 的错误和gaps,将这些scaffolds 定位到染色体上,从而得到高质量的全基因组序列。
自基因组测序以来的组装里程碑
第三代测序技术又称为单分子测序技术,主要是指pacific biosciences公司推出的single molecular real time(SMRT)测序技术(通过荧光辨别)和Oxford nanopore technologies公司推出的纳米孔测序技术(通过电流辨别)。测序时,DNA不需要PCR扩增过程,即可实现对每一条DNA分子的单独测序。
Pacbio测序平台下SMRT测序技术的两种模式:
Standard sequencing for continuous long reads(CLR)超长度长测序:存在随即错误;
Circular consensus sequen ...
Analysis_of_transcriptome
以下机数据为基础,跑通的转录组分析流程
一、部分软件安装利用conda安装trimmomatic、fastqc、hisat2、samtools等软件
HTSeq的安装需要在python2.7环境下(方法如下):
12345678wget https://pypi.python.org/packages/source/H/HTSeq/HTSeq-0.6.1p1.tar.gztar -zxvf HTSeq-0.6.1p1.tar.gzcd HTSeq-0.6.1p1/python setup.py buildpython setup.py installvi ~/.bashrcexport PATH="$PATH:/(省略)/software/HTSeq-0.6.1p1/build/scripts-2.7/htseq-count"source ~/.bashrc
注:安装的htseq-count在HTSeq-0.6.1p1/build/scripts-2.7/目录下。
注:安装的htseq-count在HTSeq-0.6.1p1&# ...
利用python快速得到两条同源序列的差异位点
准备(windows为例):安装python、pycharm
输入文件: fasta格式,包括两条序列;
输出文件包括位置及两个差异位点
12345678910111213141516# -*- coding = utf-8 -*-# @Time : 2021/5/28 23:22# @Author : wutz# @File : call_divergent_sites.py# @Software : PyCharmfasfile = open("input.fas")outfile = open("divergent-sites.txt","w")lines = fasfile.readlines()a = lines[1].strip()b = lines[3].strip()len = len(a)i = 0while i < len: if a[i] != b[i] and a[i] != "-" and b[i] != "-" and a[i] != & ...
如何利用query序列在本地匹配测序原始reads
利用转录组或重测序数据原始reads匹配到目的序列,以验证序列的准确性。
以RNA-Seq of Hippopotamus amphibius: adult male skin——SRR8270566为例
下载相关软件及工具:bwa、samtools、fastq
1234567891011prefetch SRR8270566 #下载SRA数据库的reads数据,另:wget -c https://sra-downloadb.be-md.ncbi.nlm.nih.gov/sos1/sra-pub-run-2/SRR8270566/SRR8270566.1 也可fastq --split-3 SRR8270566.1 #转换为fastq格式bwa index -a bwtsw hamp.fas #目的序列建立索引bwa mem -t 20 -M -R "@RG\tID:hamp\t" hamp.fas SRR8270566.1_1.fastq SRR8270566.1_2.fastq > hamp.sam #比对,生成sam文件samtools ...
shell三剑客
利用命令行快速处理文件
1、Shell里的循环与判断结构for循环:
1for i in `cat 1.txt`;do command;done
while循环:
1while [表达式];do command;done
if结构:
12345if [条件];then commandelse commandfi
2、shell三剑客(sed和awk部分参考了黑马程序员武汉中心在线教程文件2019全新Shell脚本从入门到精通教程_哔哩哔哩_bilibili)
grep语法格式:
1grep [options] "patten" filename1 filename2 filename3 #打印包含patten的行
常用options:
选项
说明
-v
反向输出
–color=auto
标记匹配颜色
-E
使用正则表达式
-o
只输出匹配部分
-c
统计包含匹配字符的行数
-n
输出匹配内容及其行号
-b
打印匹配模式位于行的位置
-l
搜索多个文件,并查找匹配文本在拿个文件中
. -r
在 ...
conda
使用conda安装软件以及环境管理
官网conda下载安装程序wget https://repo.continuum.io/miniconda/Miniconda3-4.3.21-Linux-x86_64.sh安装bash Miniconda-latest-Linux-x86_64.sh安装过程中一直yes,并且将其设置为环境变量,然后重新执行.bashrcsource ~/.bashrc添加频道conda config --add channels conda-forgeconda config --add channels r conda config --add channels bioconda
安装生信软件conda install softwarename
conda管理环境的部分命令集合:
#创建名为env_name的新环境
conda create -n env_name
#克隆原有的环境old_env_name,并命名为new_env_name
conda create -n new_env_name –clone old_env_name
#激活env ...
生命之源:海洋
Here we come ~