主题:基因组注释 相关参考:
基因组注释(一):重复序列注释 | 生信技工 (yanzhongsino.github.io)
使用RepeatModeler从头预测基因组重复序列 | BioChen 博客
【基因组注释】RepeatMasker和RepeatModeler安装、配置与运行避坑 - 简书 (jianshu.com)
基因组重复序列注释repeatmask&repeatmodeler - 知乎 (zhihu.com)
非模式生物重复序列注释 RepeatModeler2+RepeatMasker4 - 简书 (jianshu.com)
基因组注释(2)——散在重复序列注释 - 我的小破站 (phantom-aria.github.io)
使用AUGSTUS+Geneid+GeneMark+GeMoMa+GenomeThreader+Exonerate进行基因结构预测 - 知乎 (zhihu.com)
基因组结构注释软件列表 - 简书 (jianshu.com)
基因组注释(1)——串联重复序列注释 - 我的小破站 (phantom-aria.github.io)
1 重复序列注释 1.1 重复序列的主要分类 重复序列包括串联重复(tandem repeats)和散布重复(dispersed repeats)。前者按照重复的长度可划分为卫星序列(>100bp)、小卫星序列(>10bp and<100bp)和微卫星序列(SSR,<10bp),没有固定的长度划分标准。后者也叫做转座子(transposable element,TE),包括DNA转座子(DNA transposons,也叫做Ⅱ型转座子)和RNA转座子(Retrotransposon,也叫做Ⅰ型转座子、反转录转座子),其中RNA转座子包括长末端重复序列LTR、长散布核元件LINE和短散布核元件SINE。
1.2 注释软件 TRF:检测序列中串联重复序列,全称为Tandem Repeat Finder;
GMATA:分析基因组中的SSR序列;
RepeatScout:从头注释重复序列的核心组件;
RECON:从头注释重复序列的核心组件;
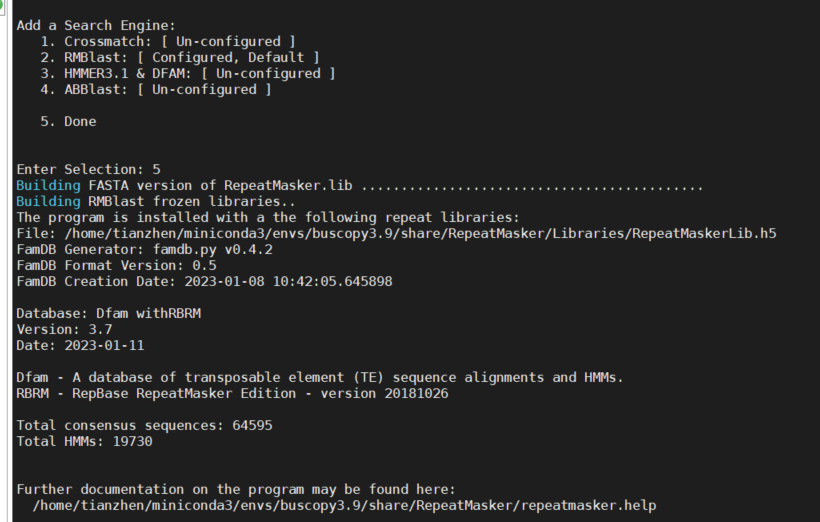
RepeatModeler:主要针对非模式物种的从头注释;RepeatMasker version 4.1.5
RepeatMasker:通过重复序列数据库(包括Dfam和Repbase等)和从头注释得到的数据库进行注释得到整合的结果;
文献中软件使用的案例:
①The rough-toothed dolphin genome provides new insights into the genetic mechanism of its rough teeth (Huang, et al., 2023)
②Chromosome-level genome assembly of hadal snailfish reveals mechanisms of deep-sea adaptation in vertebrates (Xu, et al., 2023)
③Genome assembly of the deep-sea coral Lophelia pertusa (Herrera, et al., 2022)
④The FirstDraft Genome of a Cold-Water Coral Trachythela sp. (Alcyonacea: Stolonifera: Clavulariidae) (Zhou, et al., 2020)
1.3 用于同源注释的数据库 ①Dfam:RepeatMasker自带的库,提供了大量已知的重复元件家族的信息;
②Repbase:用于存储和管理已知重复序列的数据库,在网上可以下载到2018年的版本,更新的版本则需要注册费下载;
下载并配置repbase库,进入到/home/tianzhen/miniconda3/envs/buscopy3.9/share/RepeatMasker/目录下,运行配置命令
③dc20170127-rb20170127数据库:不太了解,仅在几篇推文中看到过;
④通过RepeatModeler对自己的物种进行从头注释的数据库;
1.4 软件安装与使用 ①首先注释串联重复序列,利用GMATA和TRF两种软件。
GMATA安装非常简单,没有conda途径。下载链接https://sourceforge.net/projects/gmata/,解压后为一系列perl脚本和配置文件等,首先修改default_cfg.txt文件中参数设置,将[set]:doprimer_smt、[set]:elctPCR和[set]:mk2gff3的ModulRun = Y改为ModulRun = N,三者是设计SSR引物和模拟PCR的时候需要调用的,若设置为Y还得需要下载其他依赖。
1 2 3 4 5 6 7 8 9 perl gmata.pl -c default_cfg.txt -i /project/tianzhenWu/z189/03genome_annotation/purged.fa conda install bioconda::trf trf ../purged.fa 2 7 7 80 10 50 500 -f -d -h -r perl ~/scripts/repeat_to_gff.pl purged.fa.2.7.7.80.10.50.500.dat
结果解读:GMATA和TRF的结果具有重叠性,前者的默认参数预测了以2-10bp为单位的串联重复(SSR),后者的默认参数则会预测2000bp以下为单位的串联重复,这里我后续的分析并非以SSR为主,因此只用了TRF的结果。TRF结果的统计见推文基因组注释(1)——串联重复序列注释 - 我的小破站 (phantom-aria.github.io) ,内含脚本,写得非常详细 [点赞]。
②针对散在重复序列,结合从头注释和同源注释的思路。先利用RepeatModeler进行自我比对,建立本地数据库,然后利用RepeatMasker根据公共数据库近缘物种的重复序列和本地自建的数据库进行重复序列的从头注释。
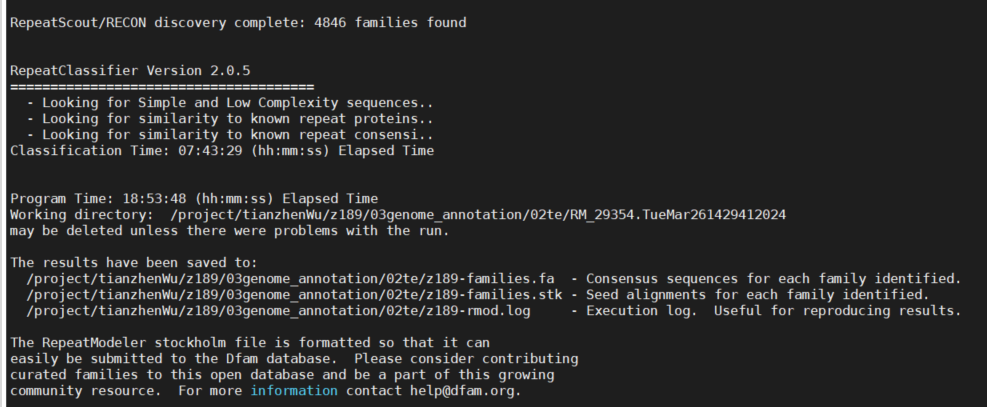
1 2 3 4 5 6 conda install repeatmodeler=2.0.5 BuildDatabase -name z189 -engine ncbi ../purged.fa RepeatModeler -pa 20 -database z189 -engine ncbi
RepeatModeler运行五轮结束(可能也会运行6轮),日志文件如下:
结果主要包括z189-families.fa、z189-families.stk和RM_29354.TueMar261429412024目录,目录下包括各种过程文件。
利用RepeatModeler的结果z189-families.fa文件运行RepeatMasker,此时运用-lib参数指定目标文件,然而当利用重复序列数据库运行RepeatMasker时,则运用-species参数指定目标物种,
1 RepeatMasker ../purged.fa -lib z189-families.fa -e ncbi -pa 40 -poly -html -gff -dir ./denove/
利用重复序列数据库近缘物种的重复序列运行RepeatMasker,先找到目标物种的近缘种是否在数据库中,然后提取近缘物种的重复序列,形成近缘物种重复序列数据库,将其与上面RepeatModeler产生的自身比对重复序列数据库合并,形成最终用于从头注释的库repeat_for_anno.fasta。
1 2 3 4 5 6 7 8 9 10 python famdb.py -i Libraries/RepeatMaskerLib.h5 lineage -ad Anthozoa python famdb.py -i Libraries/RepeatMaskerLib.h5 families -f embl -a -d Anthozoa > Anthozoa.embl buildRMLibFromEMBL.pl Anthozoa.embl > Anthozoa.fasta cat /home/tianzhen/miniconda3/envs/buscopy3.9/share/RepeatMasker/Anthozoa.fasta ../z189-families.fa > repeat_for_anno.fasta RepeatMasker -a -xsmall -nolow -norna -html -gff -dir ./xsmall_output -lib repeat_for_anno.fasta -e ncbi -pa 50 -poly ../../purged.fa
the neutral mutation rate (μ) was calculated using r8s v1.8.1
2 非编码RNA注释 2.1 非编码rRNA分类 非编码RNA (ncRNA)包括rRNA, tRNA和其他小RNA (miRNA、snRNA等)。更详细的分类可以在Rfam网站进行查询Rfam: Search Rfam 。
2.2 注释软件 ①Rnammer:一种用于预测原核生物和真核生物基因组中的RNA基因的工具,能够识别并定位16S/18S、5S和23S/28S rRNA基因序列,该软件利用隐马尔可夫模型(HMM)来识别RNA基因的保守结构特征,从而进行预测和定位。
②tRNAscan-SE:用于预测原核生物和真核生物基因组中转运RNA(tRNA)基因的工具,其结合了序列比对和结构预测的方法,能够准确地识别tRNA基因序列并预测其二级结构。tRNAscan-SE软件具有较高的准确性和灵敏度,为基因组注释、生物信息学研究和进化分析提供重要的信息。
③INFERNAL:运用Cmsan程序查找各类非编码RNA,包括miRNA、snRNA等。
2.3 用于注释的数据库 RFAM数据库是一个专门用于存储和注释非编码RNA序列的数据库Rfam: The RNA families database 。可以在这个页面Rfam: Search Rfam 查看不同非编码RNA的分类,以及通过sequence search功能来查看我们所感兴趣的序列片段是否属于某一个非编码RNA家族。
2.4 软件安装与使用 2.4.1 利用Infernal软件注释ncRNA 参考基因组注释(四):非编码RNA的注释-用Infernal软件对Rfam 12进行RNA注释 | 生信技工 (yanzhongsino.github.io)
1 conda install infernal=1.1.5
在Rfam网站Rfam: The RNA families database 下载RNA family数据库h:版本为Rfam 14.10 (November 2023, 4170 families),下载配套的clanin文件
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 wget -c https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.cm.gz wget -c https://ftp.ebi.ac.uk/pub/databases/Rfam/CURRENT/Rfam.clanin gunzip Rfam.cm.gz cmpress Rfam.cm cmscan -Z 1650 --cut_ga --rfam --nohmmonly --fmt 2 --tblout sample.tblout -o sample.result --clanin Rfam.clanin Rfam.cm ../purged.fa.masked perl infernal-tblout2gff.pl --cmscan --fmt2 sample.tblout >sample.infernal.ncRNA.gff3 awk 'BEGIN{OFS="\t";}{if(FNR==1) print "target_name\taccession\tquery_name\tquery_start\tquery_end\tstrand\tscore\tEvalue"; if(FNR>2 && $20!="=" && $0!~/^#/) print $2,$3,$4,$10,$11,$12,$17,$18; }' sample.tblout >sample.tblout.xls cat rfam.txt | awk 'BEGIN {FS=OFS="\t"}{split($3,x,";");class=x[2];print $1,$2,$3,$4,class}' > rfam_anno.txt awk 'BEGIN{OFS=FS="\t"}ARGIND==1{a[$2]=$5;}ARGIND==2{type=a[$1]; if(type=="") type="Others"; count[type]+=1;}END{for(type in count) print type, count[type];}' rfam_anno.txt sample.tblout.xls >sample.ncRNA.statistic grep "miRNA" rfam_anno.txt |cut -f1 >miRNA.tem grep -f miRNA.tem sample.tblout.xls >miRNA.txt awk '{sum += (int($5) - int($4) >= 0 ? int($5) - int($4) : int($4) - int($5)) + 1} END {print sum}' miRNA.txt
本部分详细解读请查阅生信技工的博客,非常详细且无报错基因组注释(四):非编码RNA的注释-用Infernal软件对Rfam 12进行RNA注释 | 生信技工 (yanzhongsino.github.io)
2.4.2 利用rnammer 注释rRNA rnammer软件不能通过conda进行安装,目前只允许院校研究人员通过单位邮件来申请,申请地址为RNAmmer 1.2 - DTU Health Tech - Bioinformatic Services ,提交后立即发送下载链接到邮箱,下载链接4小时内有效,下载链接如下。
1 2 3 wget -c rnammer-1.2.Unix.tar.gz tar zxf rnammer-1.2.Unix.tar.gz
安装hmmer2.3.2和perl模块
1 2 3 4 5 6 7 8 9 10 11 12 13 conda install perl-xml-simple wget http://eddylab.org/software/hmmer/hmmer-2.3.2.tar.gz tar -xf hmmer-2.3.2.tar.gz cd hmmer-2.3.2./configure make cd src;hmmsearch
在rnammer文件的目录下修改rnammer中的绝对路径
1 2 3 4 5 6 7 8 9 perl -p -i -e 's/(my \$INSTALL_PATH).*/$1 = \"\/project\/tianzhenWu\/software\/rnammer\";/' rnammer perl -p -i -e 's/^(\s+\$HMMSEARCH_BINARY).*/$1 = \"\/project\/tianzhenWu\/software\/hmmer-2.3.2\/src\/hmmsearch\";/' rnammer perl -p -i -e 's/--cpu 1//g' core-rnammer perl /project/tianzhenWu/software/rnammer/rnammer -S euk -multi -m lsu,ssu,tsu -gff rRNA.gff2 -f rRNA.fasta -h rRNA.hmmreport -xml rRNA.xml /project/tianzhenWu/z189/03genome_annotation/03ncrna/purged.fa.masked
此部分主要参考How to Install RNAmmer in Prokka - Matin Nuhamunada 、本地rnammer-1.2使用 - 简书 (jianshu.com) 和RNAmmer的安装和使用 | 陈连福的生信博客 (chenlianfu.com)
2.4.3 利用tRNAscan-SE注释tRNA 利用conda安装
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 conda install trnascan-se=2.0.12 Basic Options -E : search for eukaryotic tRNAs (default) -B : search for bacterial tRNAs -A : search for archaeal tRNAs -M <model> : search for mitochondrial tRNAs options: mammal, vert -O : search for other organellar tRNAs -G : use general tRNA model (cytoslic tRNAs from all 3 domains included) -L : search using the legacy method (tRNAscan, EufindtRNA, and COVE) use with -E, -B, -A, -O, or -G -I : search using Infernal (default) use with -E, -B, -A, -O, or -G -o <file> : save final results in <file> -f <file> : save tRNA secondary structures to <file> -m <file> : save statistics summary for run in <file> (speed, -H : show both primary and secondary structure components to covariance model bit scores -q : quiet mode (credits & run option selections suppressed) -h : print full list (long) of available options tRNAscan-SE -E -o tRNA.out -f tRNA.ss -m tRNA.stats ../purged.fa.masked
3 编码基因注释 3.1 基因结构预测的方案 一般包括三部分:①利用近缘物种的蛋白进行同源注释;②利用转录组或isoform测序数据进行基因模型预测;③通过训练已有的数据来从头预测基因组的基因结构。
3.2 参考文章示例 ①A draft genome assembly of reef-building octocoral Heliopora coerulea (Ip, et al., 2023)
②Penaeid shrimp genome provides insights into benthic adaptation and frequent molting (Zhang, et al., 2019)
③Chromosome-level genome assembly of the silver pomfret Pampus argenteus (Wei, et al., 2024)
3.3 基因结构预测的软件 这部分内容在Mr_我爱读文献 的简书中基因组结构注释软件列表 - 简书 (jianshu.com) 进行了详细罗列,不再赘述。
3.4 软件安装及其使用 3.4.1基于转录组数据注释 策略一:HISAT2 + StringTie + TransDecoder
参考基因结构预测 |TransDecoder和PASA基于转录组预测及转录本去冗余 - 知乎 (zhihu.com) 和基因结构注释(3):转录组注释 - 生信学习 | Zhou Xiaozhao = 小钊の笔记 = 前天是小兔子,昨天是小鹿,今天是你
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 conda install fastp=0.23.4 conda install hisat2=2.2.1 conda install samtools=1.19.2 conda install stringtie=2.2.1 fastp -i Unknown_BD751-01T0002_good_1.fq.gz -o output.R1.fq -I Unknown_BD751-01T0002_good_2.fq.gz -O output.R2.fq -w 8 hisat2-build ../purged.fa.masked z189.genome.index $ hisat2 -p 20 --dta --no-mixed -x z189.genome.index -1 output.R1.fq -2 output.R2.fq --no-unal -S z189.sam 2>z189.summary.txt samtools view -b z189.sam -o z189.bam samtools sort z189.bam -o z189.sort.bam stringtie -p 20 -o 02out.gtf z189.sort.bam
最后利用transdecoder注释蛋白编码区域,参考使用TransDecoder寻找转录本中的编码区 - 简书 (jianshu.com)
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 conda install bioconda::transdecoder=5.5.0 conda install bioconda::diamond=2.1.9 gtf_genome_to_cdna_fasta.pl 02out.gtf ../purged.fa.masked > transcripts.fasta gtf_to_alignment_gff3.pl 02out.gtf > 02out.gff3 TransDecoder.LongOrfs -t transcripts.fasta wget ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_sprot.fasta.gz gunzip uniprot_sprot.fasta.gz diamond makedb --in uniprot_sprot.fasta --db uniprot_sprot.fasta diamond blastp -d uniprot_sprot.fasta -q transcripts.fasta.transdecoder_dir/longest_orfs.pep --evalue 1e-5 --max-target-seqs 1 > blastp.outfmt6 TransDecoder.Predict -t transcripts.fasta --retain_blastp_hits blastp.outfmt6 cdna_alignment_orf_to_genome_orf.pl transcripts.fasta.transdecoder.gff3 02out.gff3 transcripts.fasta > transcripts.fasta.transdecoder.genome.gff3
得到基于转录组数据注释的gff文件transcripts.fasta.transdecoder.genome.gff3
注:没有脚本将该文件转换为evm格式的gff文件,在整合gff文件时的策略是将其和从头注释的gff文件合并,在权重文件中标注OTHER_PREDICTION transdecoder
策略二(可注释不同转录本):trinity+pasa
首先利用trinity进行转录组的de novo组装
1 2 3 4 5 6 7 8 9 sh trinity.sh (trinity.sh内容如下) Trinity --seqType fq \ --left Unknown_BD751-01T0002_good_1.fq.gz \ --right Unknown_BD751-01T0002_good_2.fq.gz \ --CPU 20 \ --max_memory 30G
3.4.2 基于近缘物种的同源蛋白注释 ①利用miniprot软件,下载并提取近缘物种的蛋白序列,看文献中一般用5个物种左右。将蛋白map到基因组,得到gff文件,参考大神李恒的官方描述lh3/miniprot: Align proteins to genomes with splicing and frameshift (github.com) ,以下是recommended的方法
1 2 3 4 5 6 conda install miniprot=0.13 miniprot -t8 -d ref.mpi ../purged.fa.masked miniprot -t8 -I --gff ref.mpi protein.faa > out.gff3
得到基于近缘物种的同源蛋白注释gff文件out.gff3
利用脚本将该文件转换为evm格式的gff文件,脚本地址下载地址EVidenceModeler/EvmUtils/misc/miniprot_GFF_2_EVM_GFF3.py at master · EVidenceModeler/EVidenceModeler (github.com)
1 python ~/scripts/miniprot_GFF_2_EVM_GFF3.py out.gff3 > out_evm.gff3
②利用gemoma软件,需要下载近缘物种的基因组组装序列文件(fasta格式)、gff3文件和蛋白序列文件,同时也需要上面得到的转录组reads的mapping文件(sam格式)
1 2 3 4 conda create -n gemoma python=3 conda activate gemoma conda install bioconda::gemoma=1.9
该软件具有多种运行方式:
①GeMoMa GeMoMaPipeline 加参数格式,参考GeMoMa:基因同源预测软件 - 知乎 (zhihu.com)
1 GeMoMa GeMoMaPipeline threads=10 outdir=`pwd ` GeMoMa.Score=ReAlign AnnotationFinalizer.r=NO o=true t=scaffold.fa a=Genome1.gff g=Genome1.fa
②利用java命令运行,参考基因注释工具GeMoMa - 简书 (jianshu.com)
1 java -jar GeMoMa-1.9.jar CLI GeMoMaPipeline threads=4 outdir=output GeMoMa.Score=ReAlign AnnotationFinalizer.r=NO o=true t=target.fa a=ref.gff g=ref.fa
前两种方法一直报错,再尝试第三种方法
③找到pipeline.sh文件的绝对路径,比如/public/home/wutianzhen/miniconda3/pkgs/gemoma-1.9-hdfd78af_0/share/gemoma-1.9-0/pipeline.sh,再运行该sh文件,参考gemoma安装与简单用法 - 简书 (jianshu.com) 。pipeline.sh内容如下,若有转录组数据则设置8个参数,若没有转录组数据则设置前6个参数。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 #!/bin/bash jar=$(eval "ls /public/home/wutianzhen/miniconda3/pkgs/gemoma-1.9-hdfd78af_0/share/gemoma-1.9-0/GeMoMa-*.jar" ) if [ "$1 " == "tblastn" ] then tblastn=true ; else tblastn=false ; fi target_genome=$2 ref_annotation=$3 ref_genome=$4 threads=$5 out=$6 if [ $# -ne 6 ]; then lib=$7 ; reads=$8 ; echo "GeMoMa using RNA-seq data: library type=" $lib "mapped reads=" $reads time java -jar $jar CLI GeMoMaPipeline threads=$threads t=$target_genome s=own a=$ref_annotation g=$ref_genome tblastn=${tblastn} outdir=$out r=MAPPED ERE.s=$lib ERE.m=$reads ERE.c=true AnnotationFinalizer.r=NO else echo "GeMoMa without RNA-seq data" time java -jar $jar CLI GeMoMaPipeline threads=$threads t=$target_genome s=own a=$ref_annotation g=$ref_genome tblastn=${tblastn} outdir=$out AnnotationFinalizer.r=NO fi
运行sh脚本
1 sh /public/home/wutianzhen/miniconda3/pkgs/gemoma-1.9-hdfd78af_0/share/gemoma-1.9-0/pipeline.sh tblastn purged.fa.masked.fas genomic.gff dgig.fna 5 ./result FR_UNSTRANDED ../../02Transcriptome/z189.bam
第三种方法运行成功,仍遇到了两个报错,一是GeMoMa-1.9.jar的路径问题,而是target基因组的后缀问题。前两种方法的报错可能也是由于这些问题。
GeMoMa运行结束产生final_annotation.gff文件。
利用脚本将该文件转换为evm格式的gff文件,脚本地址EVidenceModeler/EvmUtils/misc/GeMoMa_gff_to_gff3.pl at master · EVidenceModeler/EVidenceModeler (github.com)
1 perl ~/scripts/GeMoMa_gff_to_gff3.pl final_annotation.gff > final_annotation_evm.gff
脚本运行时报错缺少三个模块:Gene_obj.pm、Nuc_translator.pm和Longest_orf.pm,
这些模块可以在EVidenceModeler/PerlLib/Longest_orf.pm at master · EVidenceModeler/EVidenceModeler (github.com) 下载得到
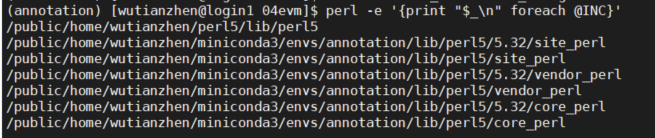
将它们放在已有的@INC目录中,这个目录通过以下命令来查询
1 perl -e '{print "$_\n" foreach @INC}'
将下载的perl模块放在/public/home/wutianzhen/miniconda3/envs/annotation/lib/perl5/core_perl/下面即可
3.4.3 从头基因结构预测 利用Augustus进行基因模型训练,训练数据可以下载近缘物种的基因组数据和gff3文件。
参考Augustus 基因从头预测 - 简书 (jianshu.com) 和AUGUSTUS - 上海交大超算平台用户手册 Documentation (sjtu.edu.cn)
建议重建一个conda环境再进行安装
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 conda install bioconda::augustus=3.5 conda install gffread=0.12.7 conda install seqkit=2.8.1 gffread genomic.gff -g GCF_004324835.1_DenGig_1.0_genomic.fna -y dgig.pep gff2gbSmallDNA.pl genomic.gff GCF_004324835.1_DenGig_1.0_genomic.fna 1000 gene.raw.gb etraining --species=generic --stopCodonExcludedFromCDS=false gene.raw.gb 2> train.err cat train.err | perl -pe 's/.*in sequence (\S+): .*/$1/' >badgenes.lst filterGenes.pl badgenes.lst gene.raw.gb > genes.gb grep '/gene' genes.gb |sort |uniq |sed 's/\/gene=//g' |sed 's/\"//g' |awk '{print $1}' >geneSet.lst seqkit grep -f geneSet.lst dgig.pep >geneSet.lst.fa makeblastdb -in geneSet.lst.fa -dbtype prot -parse_seqids -out geneSet.lst.fa blastp -db geneSet.lst.fa -query geneSet.lst.fa -out geneSet.lst.fa.blastp -evalue 1e-5 -outfmt 6 -num_threads 8 awk '$3 > 70 && $1 != $2 {print $2}' geneSet.lst.fa.blastp | sort | uniq > filtered_lines.txt awk 'NR==FNR{a[$0];next} !($0 in a)' filtered_lines.txt genomic.gff > gene_filter.gff3 gff2gbSmallDNA.pl gene_filter.gff3 GCF_004324835.1_DenGig_1.0_genomic.fna 1000 genes.gb.filter randomSplit.pl genes.gb.filter 100 new_species.pl --species=z189 etraining --species=z189 genes.gb.filter.train augustus --species=z189 genes.gb.filter.test | tee firsttest.out augustus --species=nematostella_vectensis genes.gb.filter.test | tee firsttest_nvec.out augustus --species=human genes.gb.filter.test | tee firsttest_human.out
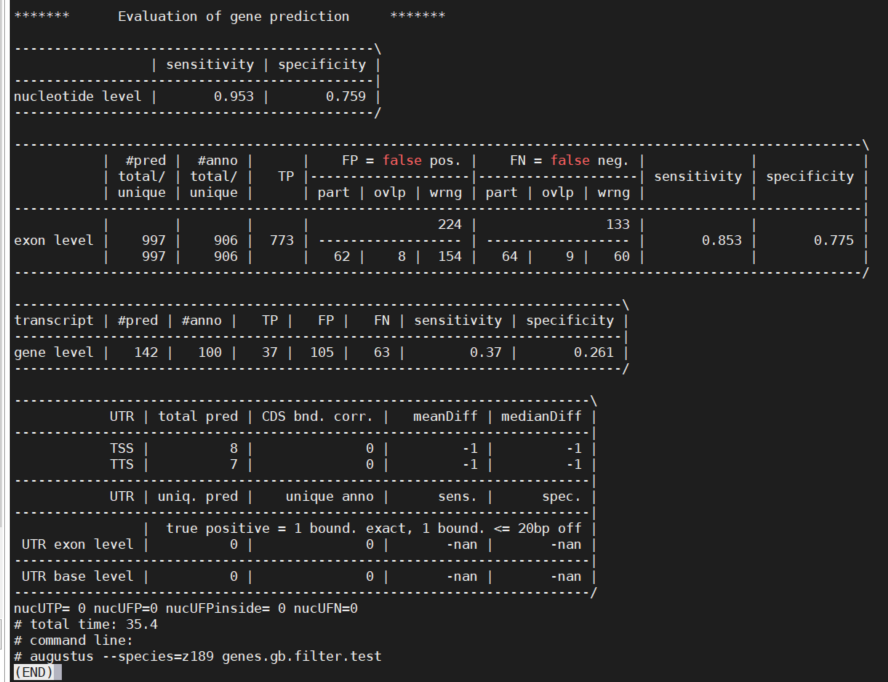
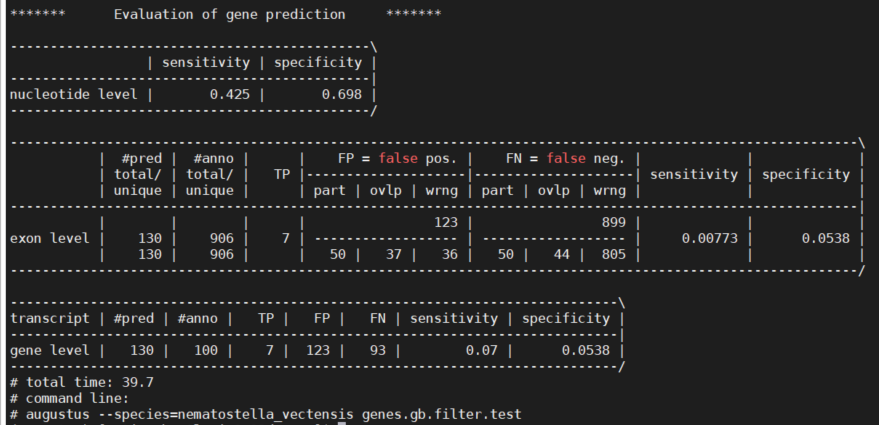
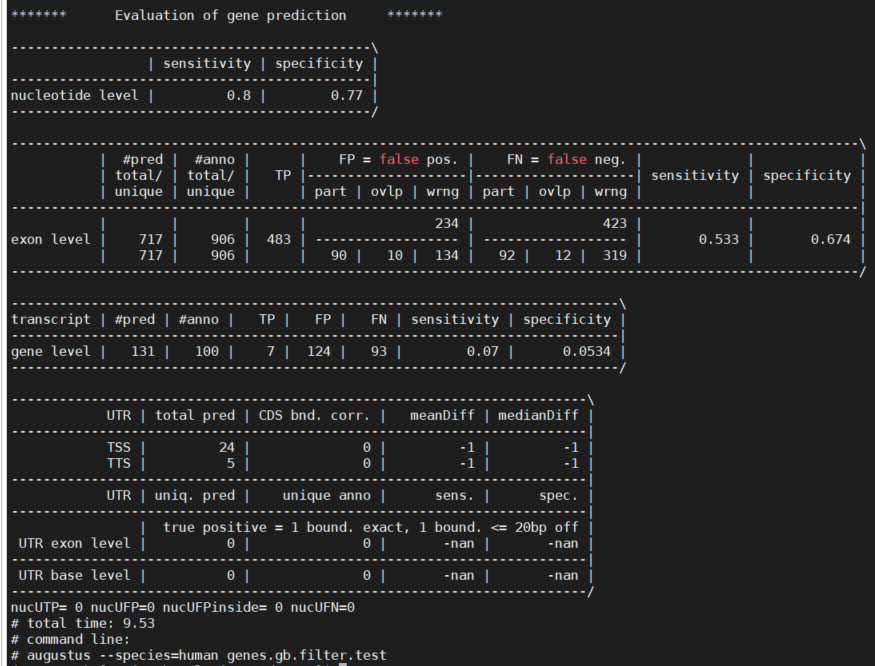
测试结果如下
近缘物种nematostella_vectensis训练情况的查看
人类human的训练情况如下
通过比较发现,训练效果优于通过近缘已训练物种的预测,但是gene level的sensitivity只有37%,不知道能不能继续下一步,还是优化训练。
其他推文说低于20%的话需要加大数据量继续优化训练,并且优化仅能提高几个百分点,因此这里初步训练的结果应该是可以拿来从头基因注释的。
接下来进行进行CRF训练,CRF: conditional random field, 进行进行CRF时,将备份/species/target species/中的所有参数。比较CRF前后预测的精确性,若升高则使用,若降低,则用上一步结果
1 2 etraining --species=z189 --CRF=1 genes.gb.filter.train augustus --species=z189 genes.gb.filter.test | tee secondtest.out.withCRF
进行基因结构预测,得到最后的gff文件augustus_z189_gene.gff
1 2 3 augustus --species=z189 --gff3=on purged.fa.masked >augustus_z189.gff perl ~/scripts/augustus_gff3_to_evm_gff3.pl augustus_z189.gff > augustus_z189_gene.gff
3.4.5 将以上不同软件的注释结果进行整合 参考EVM 对预测结果进行整合 - 简书 (jianshu.com)
1 2 3 4 wget -c https://github.com/EVidenceModeler/EVidenceModeler/archive/refs/heads/master.zip unzip master.zip cd EVidenceModeler-master/ EVidenceModeler -h
准备好三种预测的结果文件transcript_alignments.gff3,protein_alignments.gff3和gene_prediction.gff3
1 perl -p -i -e 's/^#.*//s' gene_prediction.gff3 transcript_alignments.gff3 protein_alignments.gff3
编辑注释证据的权重文件
1 2 3 4 5 6 7 8 9 vi weights.txt PROTEIN miniprot 2 PROTEIN GAF 1 PROTEIN GeMoMa 1 TRANSCRIPT transdecoder 8 ABINITIO_PREDICTION AUGUSTUS 1
分步运行程序
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 cat transcript_alignments.gff3 gene_prediction.gff3 > gene_prediction_combined.gff3 perl EVidenceModeler-master/EvmUtils/partition_EVM_inputs.pl --partition_dir ./partition --genome ../purged.fa.masked --gene_predictions gene_prediction_combined.gff3 --protein_alignments protein_alignments.gff3 --segmentSize 100000 --overlapSize 10000 --partition_listing partitions_list.out perl EVidenceModeler-master/EvmUtils/write_EVM_commands.pl --partitions partitions_list.out --genome ../purged.fa.masked --gene_predictions gene_prediction_combined.gff3 --protein_alignments protein_alignments.gff3 --output_file_name evm.out --weights `pwd `/weights.txt > commands.list" #运行evm程序 perl EVidenceModeler-master/EvmUtils/execute_EVM_commands.pl commands.list | tee evm_run.log #合并运行结果 perl EVidenceModeler-master/EvmUtils/recombine_EVM_partial_outputs.pl --partitions partitions_list.out --output_file_name evm.out #结果整理为gff3格式 perl EVidenceModeler-master/EvmUtils/convert_EVM_outputs_to_GFF3.pl --partitions partitions_list.out --output_file_name evm.out --genome ../purged.fa.masked find . -regex " .*evm.out.gff3" -exec cat {} \; | bedtools sort -i - > EVM.all.gff #过滤gff文件,过滤出长度不足50AA的序列 conda install gffread conda install bioconda::bioawk gffread EVM.all.gff -g ../purged.fa.masked -y tr_cds.fa bioawk -c fastx 'length($seq ) < 50 {print $name }' tr_cds.fa | sed 's/evm.model.//g' > short_aa_gene_list.txt #这一步骤利用别人的推送中的命令跑不通,进行了修改 grep -v -w -f short_aa_gene_list.txt EVM.all.gff > z189.gff
到这里,得到了初步地结构注释结果文件z189.gff
3.4.6 注释文件的优化与调整 Tbtools的相关插件可以优化
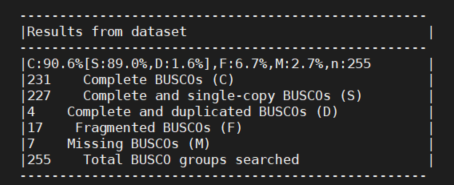
3.5 busco评估基因注释质量 1 2 3 4 5 busco_for_annotation]$ busco -i tr_cds.fa -l /project/tianzhenWu/z189/02genome_assemble/without-hic/fasta_asm/nextpolish/01_rundir/eukaryota_odb10/ -m prot -o busco_out -c 40 busco -i tr_cds.fa -l /project/tianzhenWu/z189/02genome_assemble/without-hic/fasta_asm/nextpolish/01_rundir/metazoa_odb10/ -m prot -o busco_out_m -c 40
利用真核生物库评估结果如下:
3.6 对基因特征进行可视化,包括基因长度、基因间长度、外显子个数等 玩转基因组 | 利用R语言统计外显子数量和基因长度并可视化 - 知乎 (zhihu.com)
统计注释出的基因数目,基因长度,外显子和CDS长度 - 简书 (jianshu.com)
#将gff转换为gtf文件
1 gffread z189.gff -T -o z189.gtf
1 2 3 4 5 6 7 8 9 10 11 \ cat maker_rnd3.gff | awk '{ if ($3 == "gene") print $0 }' | awk '{ sum += ($5 - $4) } END { print sum, NR, sum / NR }' 308328514 42256 7296.68 \ cat z189.gff | awk '{ if ($3 == "exon") print $0 }' | awk '{ sum += ($5 - $4) } END { print sum, NR, sum / NR }' 50327093 198915 253.008
4 基因功能注释 常用数据据库:NR,Uniprot (Swiss-Prot, TrEMBL), eggNOG, KOG, KEGG, Go,Pfam等,参考基因组注释(6)——在线版eggNOG-mapper注释功能基因 - 我的小破站 (phantom-aria.github.io)
4.1 利用NR动物数据库注释 下载nr数据库,参考创建NR子库以及从NR库提取特定物种分类的序列 | KeepNotes blog (bioinfo-scrounger.com) 和2022-09-03 NR 动物数据库构建方法2 - 简书 (jianshu.com)
1 2 3 conda install -y -c hcc aspera-cli conda install -y -c bioconda sra-tools ascp -v -k 1 -T -l 200m -i /public/home/wutianzhen/miniconda3/envs/download/etc/asperaweb_id_dsa.openssh anonftp@ftp.ncbi.nlm.nih.gov:/blast/db/FASTA/nr.gz ./
下载其他分类相关库,然后提取6073 刺胞动物子库
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 conda install -c bioconda taxonkit conda install -c bioconda csvtk wget -c ftp://ftp.ncbi.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz wget -c https://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz tar -zxvf taxdump.tar.gz cp names.dmp ~/.taxonkit cp nodes.dmp ~/.taxonkit taxonkit list --ids 6073 --indent "" > cnidaria.taxid.txt wc -l cnidaria.taxid.txt zcat prot.accession2taxid.gz |csvtk -t grep -f taxid -P cnidaria.taxid.txt |csvtk -t cut -f accession.version > cnidaria.taxid.acc.txt seqkit grep -f cnidaria.taxid.acc.txt nr -o cnidaria.fas diamond makedb --in cnidaria.fas -d cnidaria_diamond.dmnd diamond blastp --db cnidaria_diamond.dmnd --query z189.pep.fa --out nr.tab --outfmt 6 --sensitive --max-target-seqs 1 --evalue 1e-5 --id 30 --block-size 20 --index-chunks 1
4.2 利用eggNOG数据库进行GO、KEGG、Pfam注释 网页版注释eggNOG-mapper (embl.de) ,操作简单,修改e值为1e-5
4.3 利用swissprot数据库进行注释 1 2 3 4 5 wget ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_sprot.fasta.gz ./ gzip -d uniprot_sprot.fasta.gz diamond makedb --in uniprot_sprot.fasta -d uniprot_diamond.dmnd diamond blastp --db uniprot_diamond.dmnd --query ../05function_annotation/z189.pep.fa --out swissprot.tab --outfmt 6 --sensitive --max-target-seqs 1 --evalue 1e-5 --id 30 --block-size 20 --index-chunks 1
4.4 利用KOG数据库进行注释 1 2 3 4 5 6 7 wget https://ftp.ncbi.nih.gov/pub/COG/KOG/fun.txt wget https://ftp.ncbi.nih.gov/pub/COG/KOG/kog wget https://ftp.ncbi.nih.gov/pub/COG/KOG/kyva wget https://ftp.ncbi.nih.gov/pub/COG/KOG/twog diamond makedb --in kyva.fas -d kog_diamond.dmnd diamond blastp --db kog_diamond.dmnd --query ../05function_annotation/z189.pep.fa --out kog.tab --outfmt 6 --sensitive --max-target-seqs 1 --evalue 1e-5 --id 30 --block-size 20 --index-chunks 1
利用excel整理结果。
制作VN图:jvenn (inrae.fr) ,最多可以做6组。