利用TBtools绘制简单的共线性图
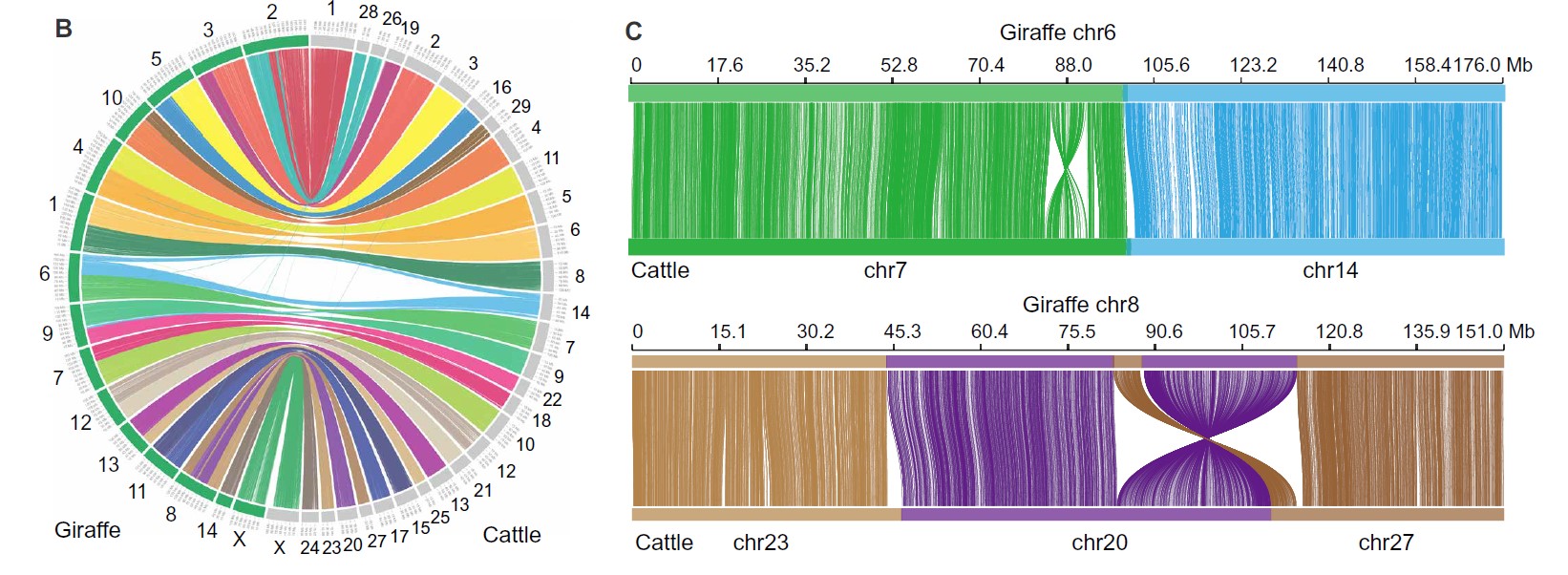
学习目标:长颈鹿基因组文章中的基因组共线性关系图。
首先测试一下TBtools中在Graphics下面Advanced Circos工具。
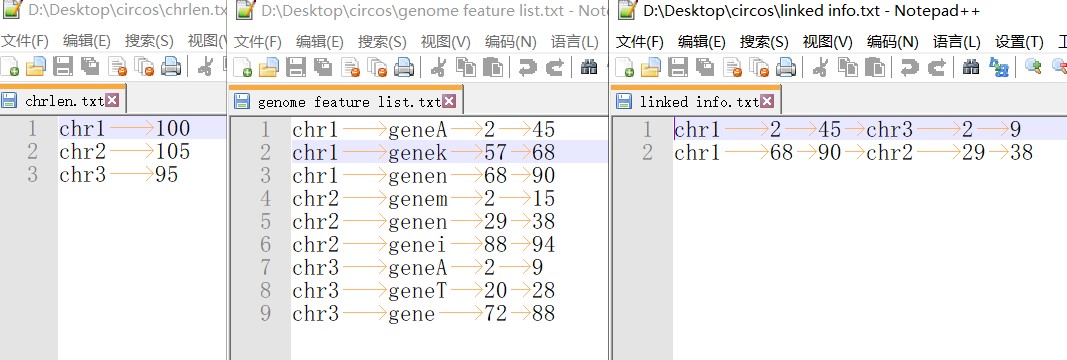
该工具下的三个输入框分别输入三个文件,文件中每列以制表符分隔开来。

①染色体的名字及其长度,共两列;
②染色体上所有元件特征的位置信息,分别为染色体名称、序列元件名称、序列原件的起始位置和序列原件的终止位置,共四列;
③同一元件在不同染色体的位置信息,分别为染色体1的名称、元件在染色体1的起始位置、染色体1的终止位置、染色体2的名称、元件在染色体2的起始位置、染色体2的终止位置。
为了方便测试,简单编辑了三个文件,格式如下
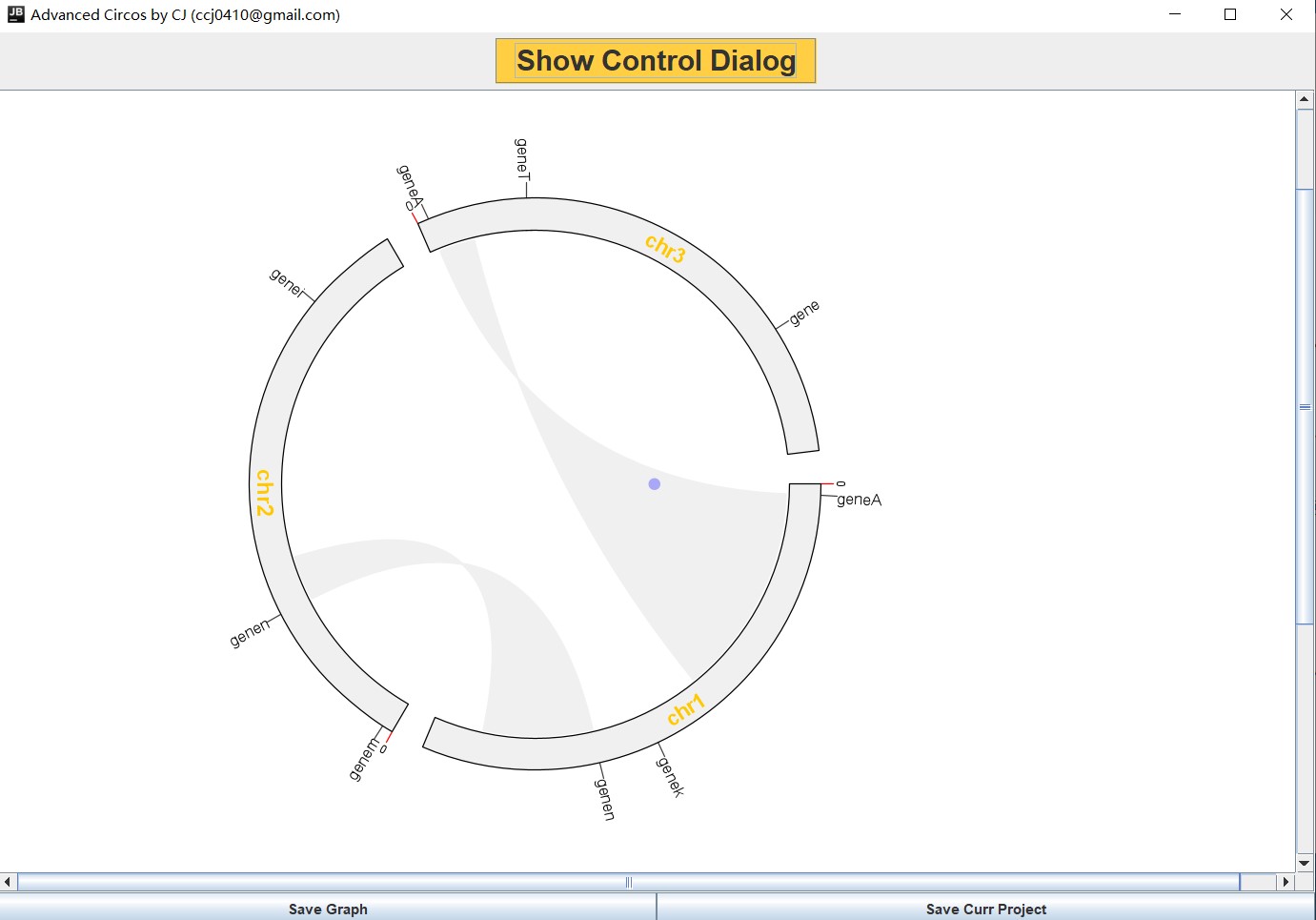
将这三个文件分别拖入输入框后,点击绘图,得到以下结果,表明没有软件报错,运行环境正常,如果用基因组大数据来绘图时,若文件格式正确,应该不会出问题。
接下来是如何在公共数据库中获取基因组数据,以及如何进行数据转换,得到circos工具需要的格式。
以多孔鹿角珊瑚(Acropora millepora)基因组为例,该物种基因组是目前珊瑚虫纲为数不多的组装为染色体水平的基因组,包括已装配的14个染色体和为装配的部分。
NCBI下载GFF(general feature format)文件后,发现14个染色体的长度在以“##sequence-region NC_”开头的行中,所以可以利用此特征进行grep得到染色体长度信息,这样就可以轻松准备好第一个文件。
1 | grep "##sequence-region NC_" GCF_013753865.1_Amil_v2.1_genomic.gff |awk '{print $2,$4}' OFS="\t" > coral-chrlen.txt |
第二个文件需要染色体上所有元件特征的位置信息,基因组上的元件一般包括gene、exon、mRNA等,一般取gene进行可视化,那么需要将GFF文件中装配在染色体上的并且第三列为gene的行提取出来。也就是提出”NC_xxxx.x Gnomon gene“格式的行,因此可以得到染色体上所有基因的所有信息。
1 | grep "NC_" GCF_013753865.1_Amil_v2.1_genomic.gff |grep $'\t'Gnomon$'\t'gene$'\t' > chr-gene.txt |
根据文件特征,利用awk和grep提取第二个文件的信息。
1 | awk -F'[\t;]' -v OFS="\t" '{print $1,$13,$4,$5}' chr-gene.txt | sed 's/gene=//g' > coral-genome-feature-list.txt |
此处得到的文件中,存在极少部分的行,其第二列并不是基因名,这是GFF文件中这一行与其他行的格式不一样导致的,在本测试中有97行出现错误,即基因名字为”gbkey=Gene“,考虑到这些基因不足总基因数的0.5%,选择直接去除这些行。
1 | sed -i '/gbkey=Gene/d' coral-genome-feature-list.txt #删除带有gbkey=Gene的行 |
下面制作第三个文件
1 | awk '{print $2}' coral-genome-feature-list.txt > all-genes-list.txt #将所有基因提取出来 |
最后将得到的三个文件导入到TBtools软件中,绘图。
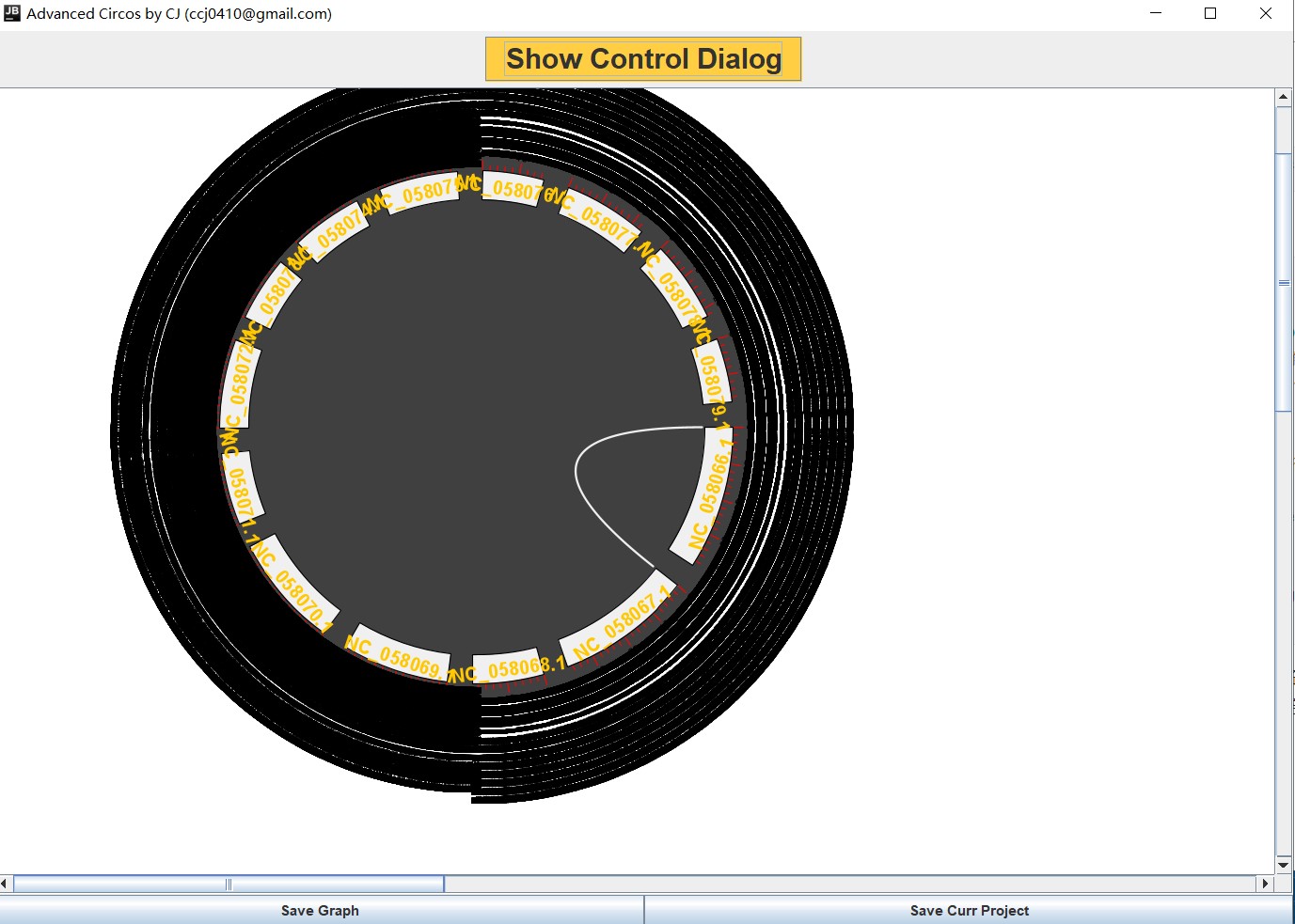
由于基因太多,基因名展示在图上重叠在一起一片漆黑,调整图片位置都卡好久,但是整体上看是符合预期的。因此,此方法适合选择部分基因来展示,不适合所有基因都进行共线性展示的情况。
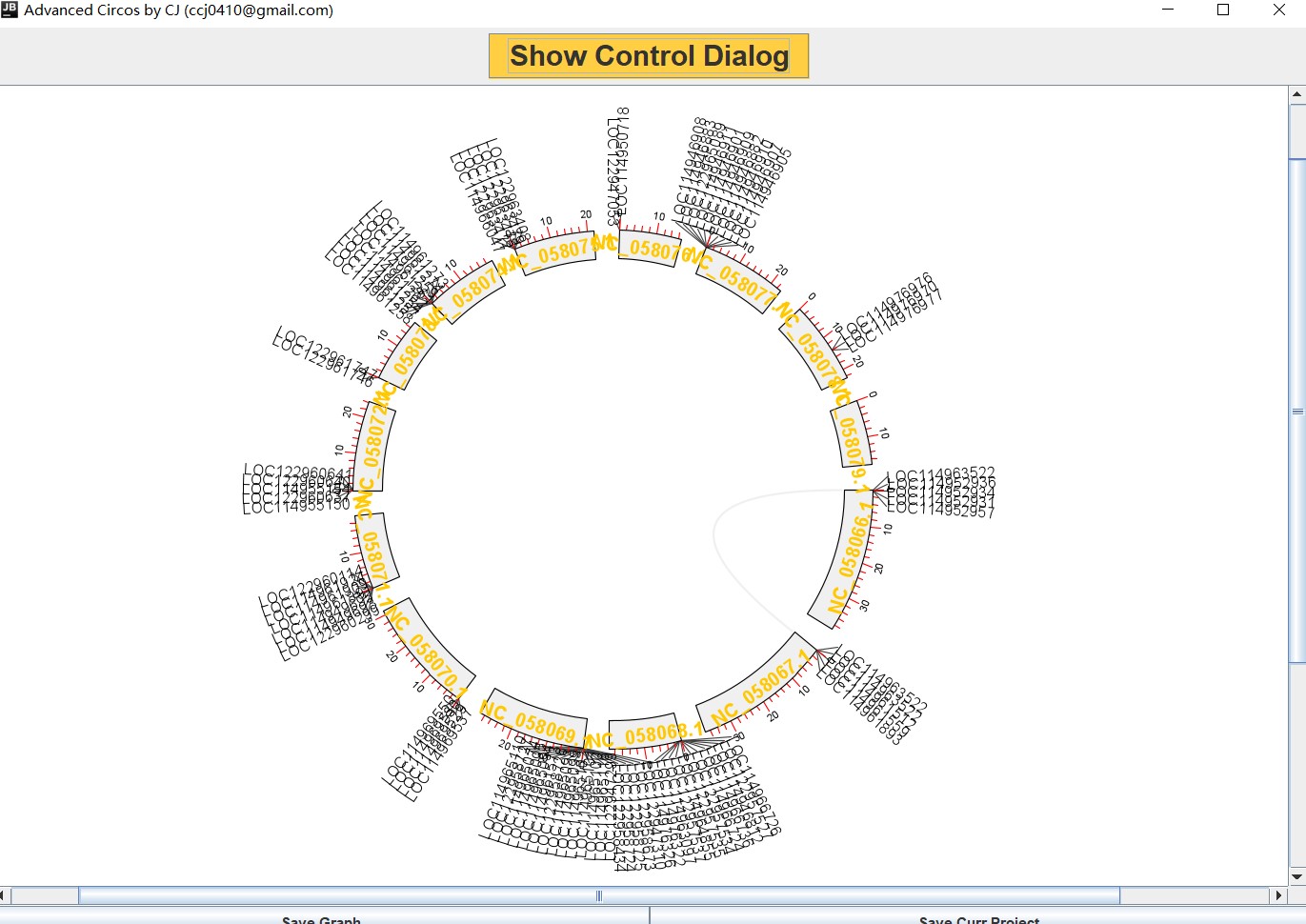
为了方便展示,并美化图,将genome-feature-list文件简化,得到以下。
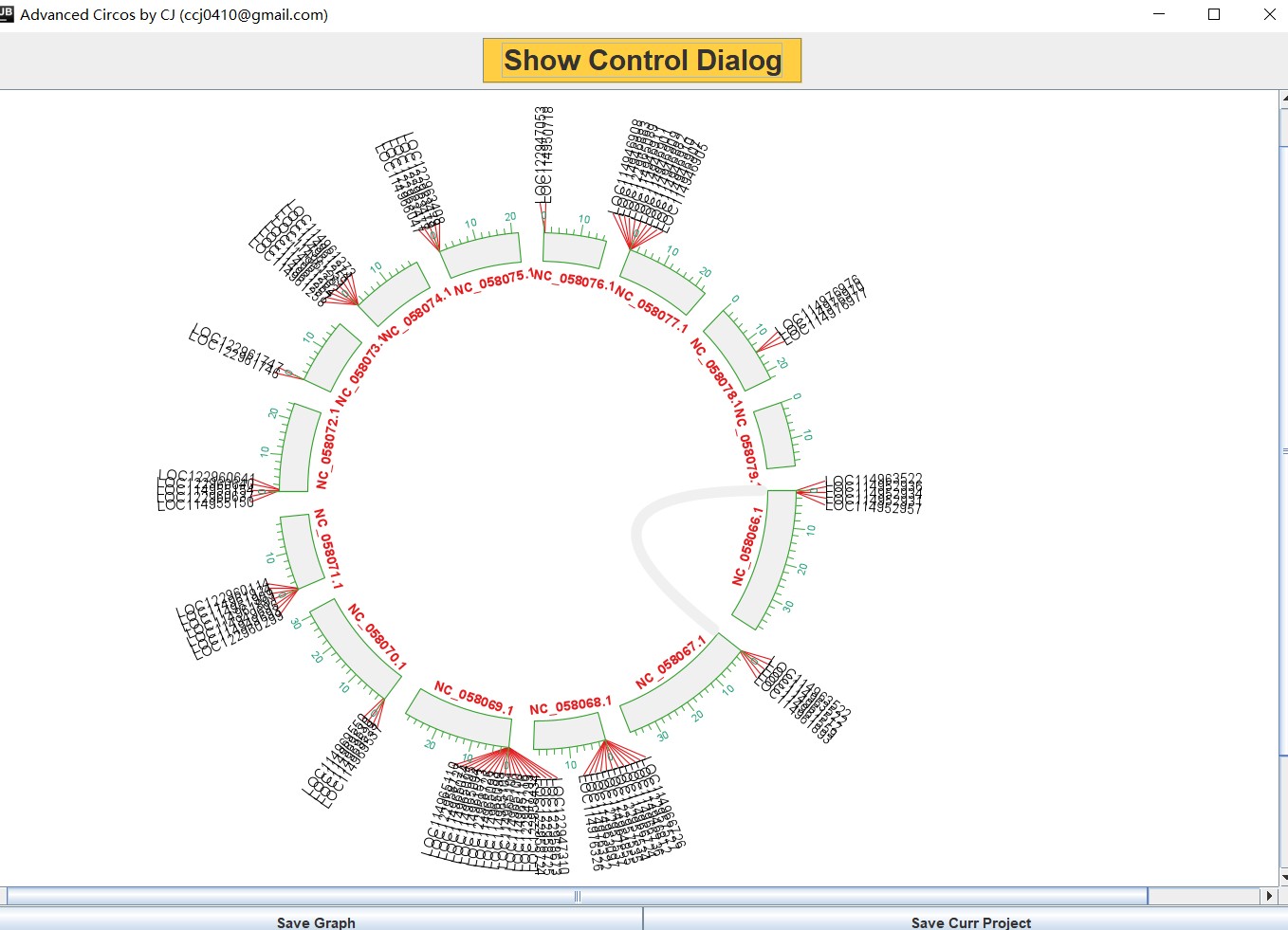
进一步调整颜色得到最终的circos图,根据自己的需要调整图片各元素的颜色和位置。
为什么做circos图?
展示基因组各种元件的信息,比如新测基因组与已测基因组的共线性情况;
表示基因的复制情况等;
……
虽然TBtools不如circos软件的命令行模式灵活,但该工具为非编程用户提供了极大的便利,值得学习。
若绘制以下复杂的基因组信息图,建议使用circos命令行模式的软件。
1 | 计算染色体长度 |
傻瓜式利用TBtools绘制两基因组的共线性关系图
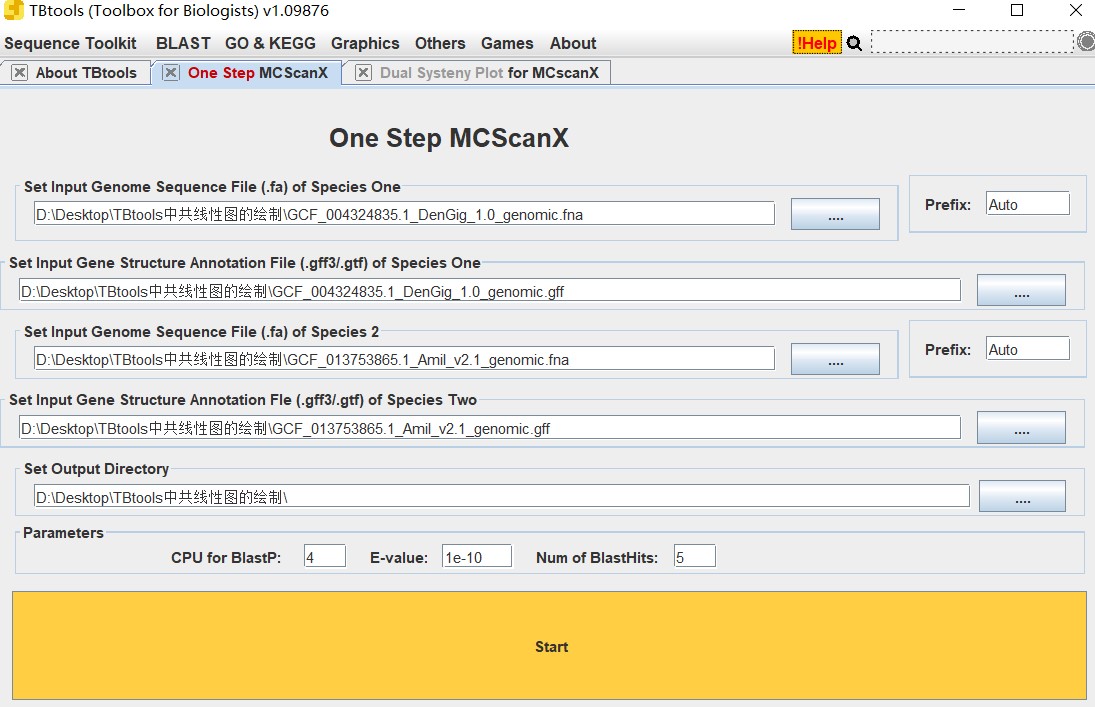
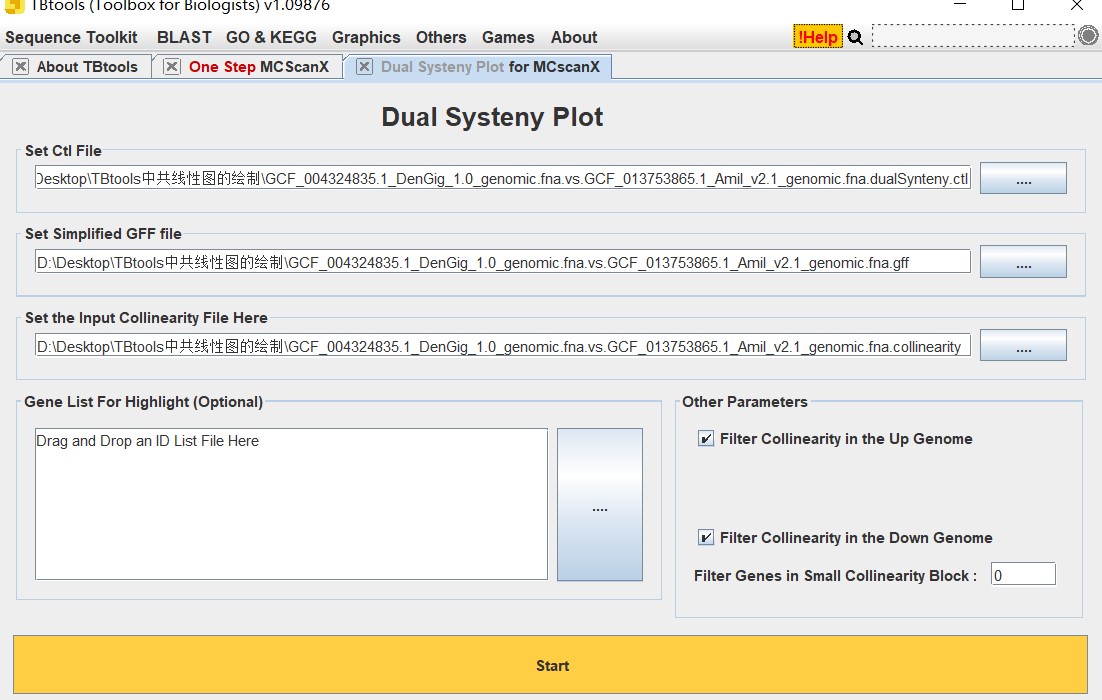
要想制作推文最初的C图,可以使用Graphics中的Comparative genomecs下的两个工具,即One Step MCScanX和Dual Systeny Plot for MCScanX,前者用于处理绘图所需的正确格式的文件,后者用于两个基因组关系的绘图。
只需要分别准备两个物种的基因组文件和GFF文件即可,这四个文件拖入第一个工具,运行耗时比较久(半小时左右?),这样就得到第二个工具的输入文件,然后拖入点击秒出图。
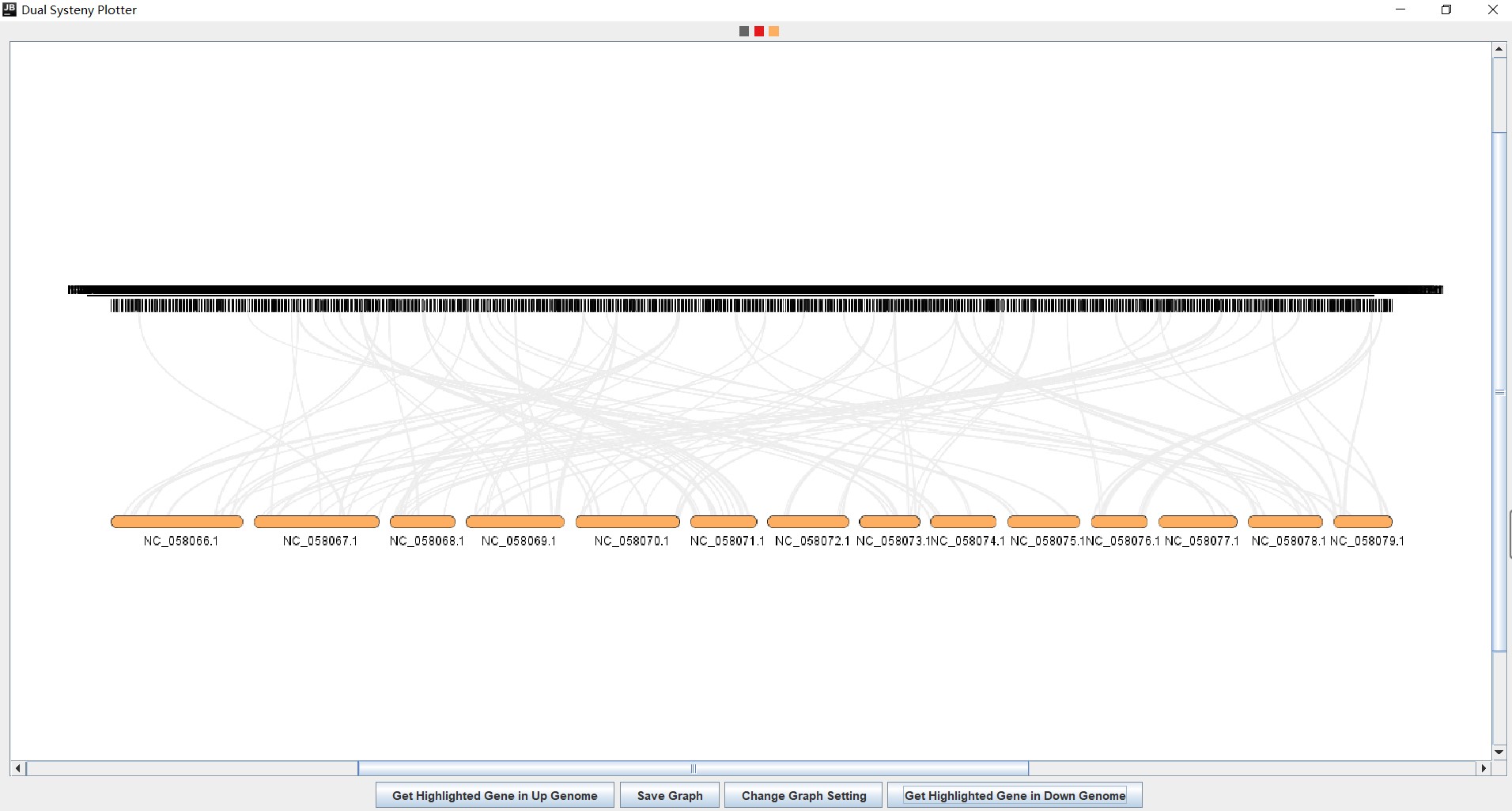
此工具不用任何思考,只需点点点就能得到这个。
注:
①最好使用两个染色体水平的基因组作图,上面的基因组仅组装到contig水平,所以看不出什么进化事件;
②可以自行编辑第一步骤得到的ctl文件,通过保留想要展示的片段,可将其他片段的名字直接删除;
③颜色可以编辑。
④NGenomeSyn是专门绘制基因组共线性的命令行软件,功能更加详细,更加灵活。
参考:
基因组Circos图绘制 - 简书 (jianshu.com)
TBtools绘制Circos图小攻略 - 百度文库 (baidu.com)