转录组测序研究进展
转录组(Transcriptome)是指特定细胞或组织中全部转录产物,包括信使RNA,核糖体RNA、转运RNA 以及非编码RNA。
1 转录组学是什么?
转录组学是从整体转录水平系统研究基因转录图谱并揭示复杂生物学通路和性状调控网络分子机制的学科。
2 转录组测序是什么?
转录组测序(RNA-seq)就是利用高通量测序技术将细胞或组织中全部或部分mRNA、small RNA和no-coding RNA 进行测序分析的技术
3 可以用来解决什么问题?
①检测与现有基因组序列相对应的转录本;②能发现和定量新的转录本;③基因转录的物种特异性和时空差异;④探究非编码RNA的调控机制;⑤单细胞转录组解析不同类型细胞的基因表达谱或空间分布情况。
怎么理解转录本与基因之间的关系呢?
研究一个基因时应该首先确定研究该基因的哪个转录本
转录本其实就是基因通过转录形成的一种或多种可供编码蛋白质的成熟的mRNA
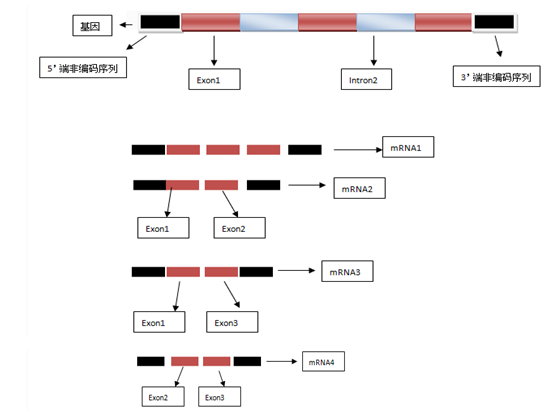
4 转录组测序的一般流程和测序内容?
4.1 mRN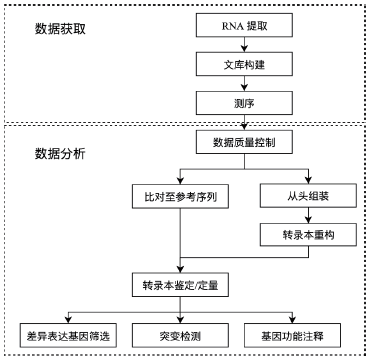 A测序
A测序
利用mRNA 在3’ 端具有poly-A 的结构特点,富集出特定组织或细胞在特定时空条件下转录出来的不含内含子序列的mRNA 分子,反转录成cDNA 建库测序。
4.2 small RNA测序
Small RNA 是指长度在20-50 nt 的RNA 分子,包括miRNA、siRNA、snoRNA 和piRNA 等, 通过参与mRNA 降解、抑制翻译过程、促进异染色质形成和DNA 表观修饰等多种途径来调控生物学过程。根据small RNA 的5’ 端磷酸基和3’ 端羟基的结构特点,链接测序接头并筛选small RNA 测序文库进行测序。miRNA 在物种间的生物学功能较为保守,是small RNA 测序研究中的重点。
4.3 lncRNA测序
长链非编码RNA(lncRNA) 是一类长度在200 nt 以上、无编码蛋白质功能的RNA 分子,往往具有很强的物种、组织特异性。部分lncRNA 位于基因的增强子区域,通过自身的转录而实现增强子的功能。lncRNA 调控方式多样且广泛存在于各类动植物细胞中,可以通过参与染色体结构形成以及与转录因子、蛋白质、RNA 前体、miRNA 结合等多种方式调节各类生物学分子的功能。部分lncRNA含有ploy-A 尾结构,因而在mRNA 的测序结果中往往包含部分lncRNA 序列信息。目前对于lncRNA 的研究,以寻找差异表达的lncRNA 分子入手,主要依据lncRNA 与关键编码基因的位置关系,进一步预测两者之间的调控关系。
4.4 circRNA测序
环状RNA(circRNA)具有特殊的稳定性良好的成环结构,不容易被RNA 酶降解,被认为在生物体内可以长效行使转录调控功能。同一段基因组序列可能会产生多种类型的circRNA 分子,外显子和内含子的不同剪切组合使得circRNA 可能包含多个外显子或内含子序列。circRNA 具有吸附miRNA分子的“海绵”作用,介入miRNA 对mRNA 的调控过程。
4.5 全转录测序 Whole transcriptome sequencing
全转录组测序(Whole transcriptome sequencing)能够测定样本中的全部完整的转录本,主要包括mRNA 和非编码RNA(lncRNA,circRNA和miRNA)。全转录本测序与常规RNA-seq 的区别主要是建库方式的不同。全转录组测序在建库过程中需分别建立2 个文库(mRNA+lncRNA+circRNA文库和miRNA 文库)或3 个文库(mRNA+lncRNA文库、circRNA 文库和miRNA 文库)。通过全转录组数据,不仅可以获得全部类型转录本的表达图谱,在此基础之上,对不同RNA 分子进行鉴定和注释,分析其编码蛋白和调控功能,并对RNA 分子之间的互作调控网络进行分析,从整体上全面系统的分析特定细胞在特定时空下的生物学特征。
4.6 单细胞转录组测序 scRNA-seq
单细胞转录组测序技术是在单细胞水平研究整个转录组的技术,用于评估单个细胞间基因表达的差异,能避免细胞类型混杂而引入的假阴性结果,有可能识别出无法通过混合细胞检测到的罕见的细胞群体。
单细胞分离是scRNA-seq 的关键步骤,主要通过连续稀释、显微操作分离、荧光激活细胞分选(Fluorescence-activated cell sorting,FACS)和微流控分离(Microfluidic technology)等技术实现。
5 构建文库的策略
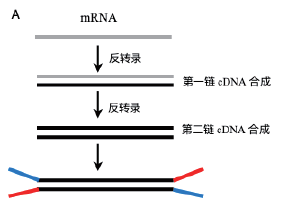
5.1 非链特异性文库(Non-strand-specific library)
RNA 逆转录成双链cDNA,随机加上接头、不区分RNA 的链的信息的文库。测序时以双链cDNA 进行测序,无法区分mRNA 的转录方向。
5.2 链特异性文库(Strand-specific library)
以化学修饰标记一条链,比如通过重硫酸盐处理RNA 分子,或者在第二链cDNA 合成时引入dUTP,然后降解含有U 的链;
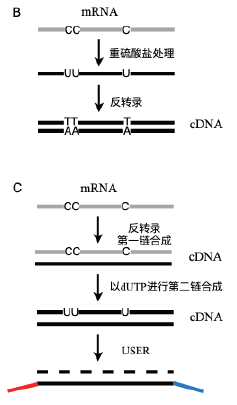
以不同接头连接RNA 分子或合成cDNA 链的5’ 和3’ 末端,来区分正反义链;
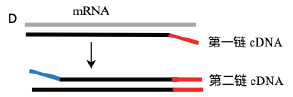
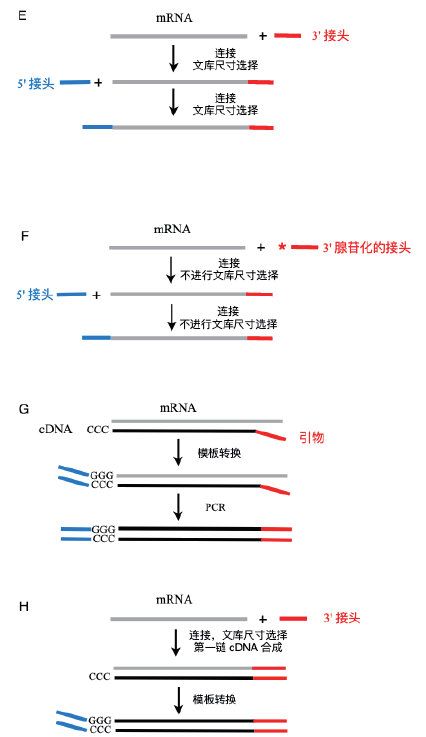

经科研人员测评,C和E两种构建文库方法效果较好。
6 数据处理流程
用于比较不同组别之间基因水平或转录本水平的定量差异时,其分析基本流程包括以下:
6.1 原始数据预处理
常用质控软件包括:Trimmomatic、RSeQC、FASTX、Trim Galore等,QC 后得到的数据称为clean data,用于后续分析。
6.2 reads比对
应用于转录组数据的比对软件, 常用的有bowtie、bowtie2、STAR、HISAT/HISAT2等,BWA软件的比对算法被认为对于分割比对不敏感,因而不适合用于RNA 序列与含有内含子序列的基因组序列之间的比对。
6.3 转录本组装
转录本组装就是将测序数据组装成转录本。对于有参考基因组的物种,根据转录组比对后的结果,明确外显子之间的连接方式,从而构建出转录本的结构。常用工具包括Cufflinks 和Scripture。
对于无参考基因组序列的转录组数据,为了获得完整的转录本序列,需要对RNA 测序得到的短reads进行从头组装。常用工具包括Trinity、TransAbySS和Velvet等。以Trinity 组装小鼠的转录组数据时,为了保证组装效果,至少需要30× 以上覆盖度的测序reads。
6.4 转录本预测
大多数基因有多种剪接形式,且有可能产生多种转录本,从而编码产生不同的蛋白,这样有可能造成一个基因有多种功能。
对于有参考基因组和转录本参考信息的物种,转录本结构主要是根据测序得到reads 进行比对,reads 覆盖了全部的转录本序列,依靠基因组序列组装出完整的转录本信息。
对于无参考基因组的物种,需要自行组装出基因的转录本序列。得到的基因或转录本序列可以与同物种或近源物种的unigene 和EST 数据库进行比较,以判断得到的基因或转录本序列的可靠性。
6.5 转录本表达水平分析
FPKM是应用于双段测序的RNA-seq 分析中。Cufflinks、DESeq/DESeq2、EDGR等软件可用来进行表达量的确定。常用FDR 等多重检验校正的方法对比较分析的显著性进行校正。
6.6 变异检测
检测转录本上全部的SNP 和Indel等突变类型。SAMtools、BCFtools和GATK等软件可用来检测转录组中相关的变异。
以上具体流程参考Analysis_of_transcriptome | Wutianzhen (wu-tz.github.io)
7 当前转录组热点
PacBio 的单分子实时测序技术,具有读长较长的优点,能够进行全长转录组的研究,特别适合用于发现新转录本。随着单细胞分离以及单分子测序技术的发展,单细胞转录组测序技术在异质性细胞的转录组研究中具有广阔的前景。
本文大篇幅摘自综述农科院崔凯老师的《转录组测序技术的研究和应用进展》。
崔凯, 吴伟伟, 刁其玉. 转录组测序技术的研究和应用进展. 生物技术通报, 2019, 35(7): 1-9






